近日,山东大学公共卫生学院/国家健康医疗大数据研究院薛付忠教授团队在国际顶尖期刊《Nature Communications》杂志上在线发表了题为“MR-EILLS: an invariance-based Mendelian randomization method integrating multiple heterogeneous GWAS summary datasets”的因果推断方法文章。国家健康医疗大数据研究院/山东大学公共卫生学院侯蕾副研究员为本论文的独立第一作者,山东大学生物统计学系陈昊助理研究员和北京大学生物统计系主任周晓华教授为共同通讯作者。《Nature Communications》杂志是国际最具权威性的专业杂志之一,也是发表遗传统计新方法的顶级期刊之一,最新影响因子为16.6,中科院一区Top期刊,JCR分区1区,为公认的综合科学领域权威刊物。本研究的主要内容如下:
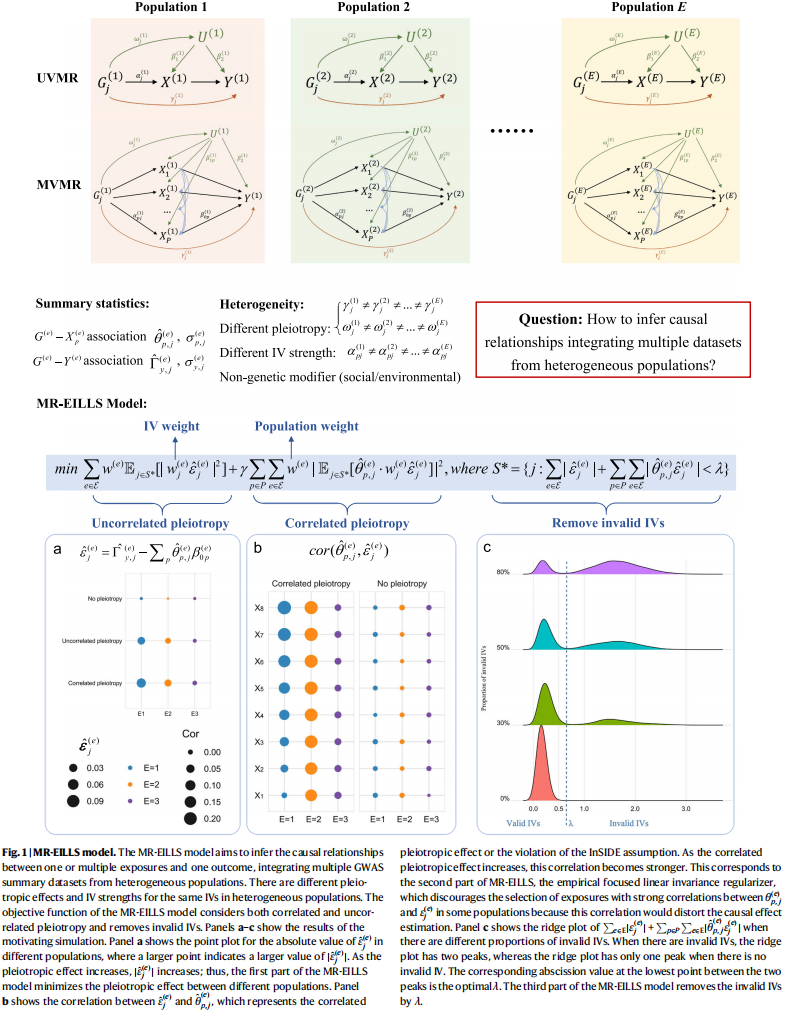
本研究针对多源异质性全基因组关联研究(GWAS)汇总数据整合中存在的遗传结构差异问题,创新性提出基于环境不变性原理的孟德尔随机化方法MR-EILLS(Environment Invariant Linear Least Squares)。该方法通过统计建模建立跨种族普适的不变因果效应估计框架,在五大种族(非洲、东亚、南亚、西班牙/拉丁裔、欧洲)的GWAS数据分析中实现重大突破:其核心优势在于同时支持单暴露及多暴露场景,可处理违反可交换性假设与排他性约束的无效工具变量,并通过理论证明确保渐近无偏性与统计一致性,为异质数据整合提供了严格的方法学基础。研究团队通过大规模模拟实验与实证分析(涵盖11种血细胞指标与20种疾病结局的因果关系验证)证实了该方法的优越性。相较于传统逆方差加权法及随机效应模型,MR-EILLS显著提升估计精度与统计功效,且I类错误率严格控制在理论水平。在生物学机制解析层面,该方法成功验证大部分已知血细胞-疾病因果链,并发现多项跨种族一致的新关联,为多祖先人群的精准医学研究提供了可靠证据。
MR-EILLS首次从数学本质上解决了异质性与无效工具变量的耦合干扰问题,建立了适用于多元族群GWAS数据的标准化因果推断范式。该模型为药物靶标验证、跨组学整合分析及种族特异性机制研究开辟了新路径。研究团队已在GitHub平台开源算法实现代码(https://github.com/hhoulei/MREILLS)并同步发布R语言软件包,推动方法学成果的快速转化应用。
文章链接:https://www.nature.com/articles/s41467-025-62823-6